复杂疾病要做wes?这些关键点你get了吗!?
样本就绪!经费就绪!相关销售合同准备就绪!马上开始实验吧!
小编忍不住用尽全力喊出:且慢!!!!
尽管全外显子测序(whole genome sequencing)应用已经到了白热化的阶段,但WES技术应用于复杂疾病研究,还是有很多关键的细节要考虑。那么,这些WES实验过程的关键点,你真的get到了吗?
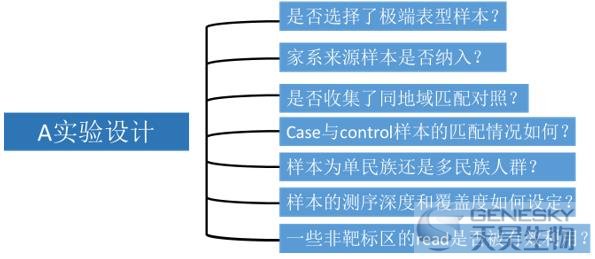
A:实验设计中的关键点
l 是否选择了极端表型样本?
极端表型样本,往往会大大提高WES研究检测致病基因的效力。对于数量性状来说,极端表型意味着在排除其它临床疾病因素下,患者表现出的异常表型,如极高的身高,血压或血糖等;对于二分类性状,极端性状意味着对于患者本人来说表现出的不寻常表型(如非常年轻的心梗患者)。 l 家系来源样本是否纳入? 对于一些先天性疾病或者婴幼儿时期发病的疾病,往往考虑de novo突变在其中的重要作用。因此,在收集这类患者样本的时候,最好同时获取父母样本帮助判断突变的来源。 l 样本的测序深度和覆盖度如何设定? 目前,wes研究普遍要求80-95%的蛋白编码区能有20X以上的高质量测序数据。为了达到这一要求,一般要求每个样本的平均测序深度至少超过80X。 l 一些非靶标区的read是否被有效利用? 对于全外显子测序试验流程,其实在非靶标区也会有0.2-2X的一个平均测序深度。但这些序列往往被直接删去,不用做后续研究。实际上,这些数据对于判断样本的人群异质性,或者是否存在大的结构变异,都有十分重要的作用。(对于某些高要求的杂志来说,这部分数据常常被用来进行人群异质性分析)。
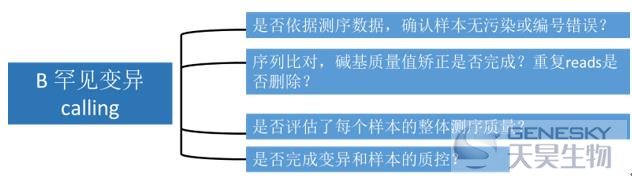
B 罕见变异calling
从测序原始数据到获得罕见变异,variant calling(识别变体)是最关键的一环。
? 是否依据测序数据,确认样本无污染或编号错误?
二代测序数据通量极大,一次实验检测的可能是多个实验室的样本,因此,在数据分析之前,判断样本的编号对应无误以及样本有无污染,是后续获得可信结果的关键。一般污染样本,会有极高的杂合度,需要重视。
? 是否评估了每个样本的整体测序质量?
基于测序数据,有很多指标能够判断测序的整体质量。比如:每个样本的变异数(一般每个样本约有1-1.25万个同义突变,0.95-1.2万个错义突变,100-200个无义或者剪切突变);各类变异的占比;转换/颠换率(对于非同义突变,一般该比率略大于2,对弈同义突变,该比率大于5)。理论上,对于一些指标非常异常的样本,往往要重点考虑是否纳入后续研究。
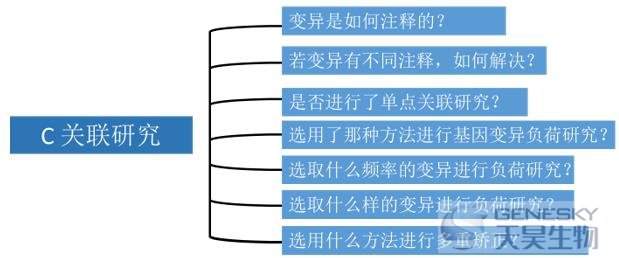
C 关联研究
? 若变异有不同注释,如何解决?
有的变异可能位于不同的转录本,而在不同转录本上的注释不同。这里一般建议只关注其在最长转录本上的注释;或者选危害程度最大的一个注释。
? 是否进行了单点关联研究?
对于wes研究项目,一般样本量远低于关联研究的样本。据统计,在99%的效力情况下检测频率为0.5% 或0.05%的变异,至少需要460 或 4,600例的样本。虽然对于wes研究,很多样本量达不到这么高,但在数据分析的时候,还是建议从单点关联研究出发。这种分析类似于GWAS,可以绘制QQ图,从而直观的反应样本的匹配情况和人群分层情况。
? 选用了那种方法进行基因变异负荷研究?
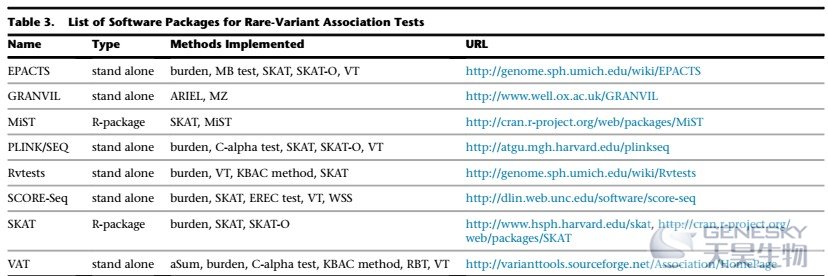
负荷研究,其实就是探讨了case样本在某个基因上的变异和control样本在某个基因上的变异是否有不同。完成这个负荷分析,有很多方法,没有统一的标准。这些方法的差异,在于2个核心问题: 是直接统计变异的个数还是对变异影响的大小进行权重后再统计;有害变异和保护变异是否综合讨论。下表展示了目前常用的多种负荷检验方法,建议采用2种以上的方法,进行负荷分析,对比结果。
? 选用什么方法进行多重矫正
对于wes研究,一般以5*10-7作为显著性的分界点。但是,这个值也是经验法则来源的。最好还是基于置换研究方法,获得多重矫正显著性值(permutation-based approaches)。
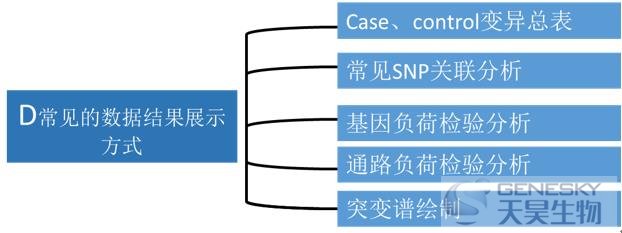
D 常见的数据结果展示方式
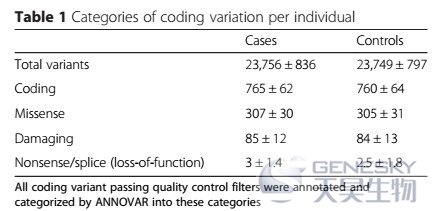
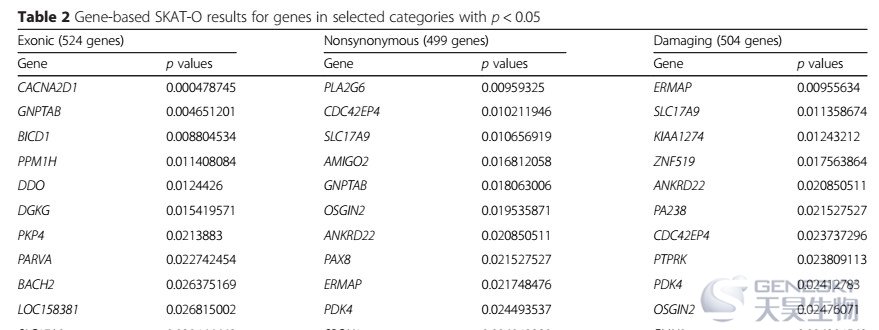
? Case、control变异总表【2】。分别统计患者和对照的各类变异数目。
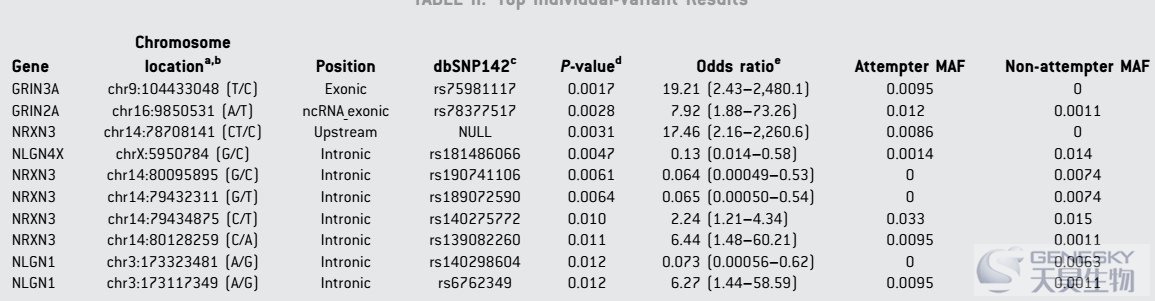
? 常见SNP关联分析【1】
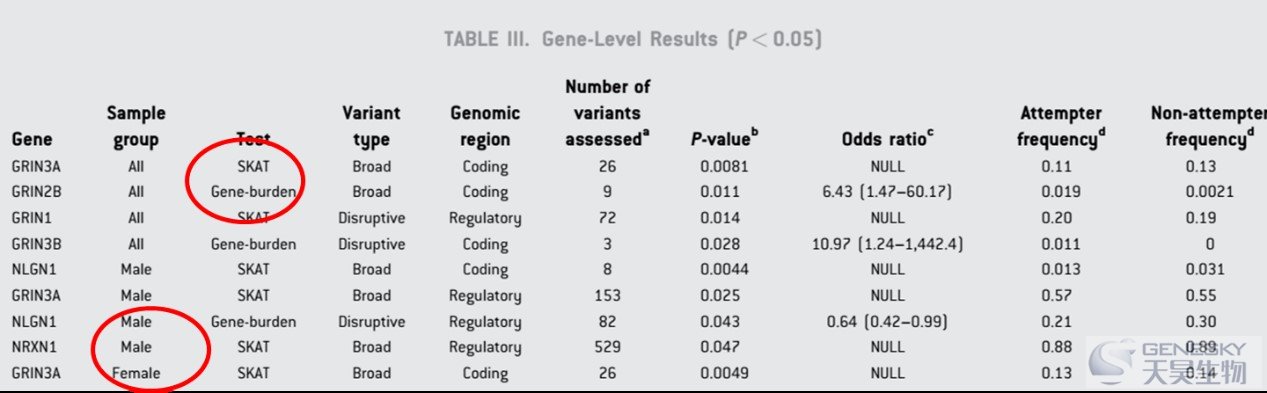
? 基因负荷检验分析;
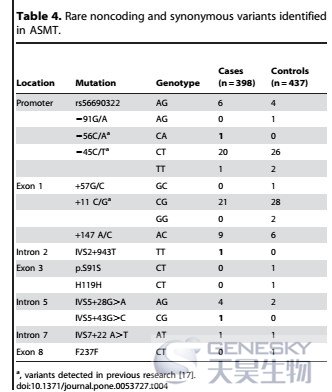
按不同位置的变异来进行负荷检验分析【4】
按不同类型的变异来进行负荷检验分析【2】
用不同的方法来进行负荷检验分析【1】
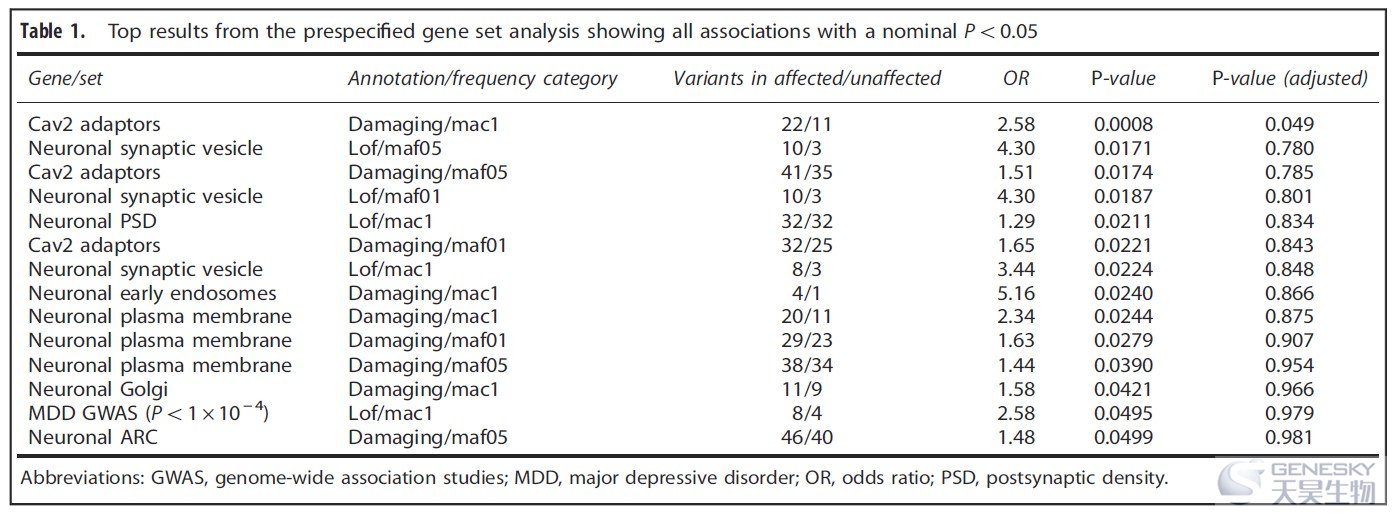
? 通路负荷检验分析;将传统的基因负荷分析进一步放大的通路层面。【3】
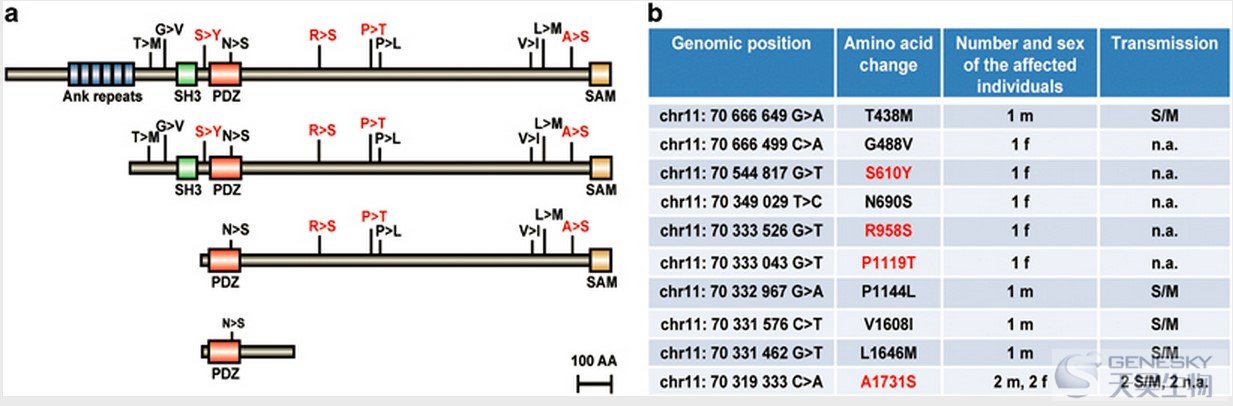
? 突变谱绘制【5】
E后续研究思路
? 阳性SNP后续的大样本关联分析验证
一项NG发表的研究,首先完成了350对样本的目的区域测序,后续在1万多例样本里检测了70个SNV的基因型,从而完成第二阶段的关联研究。【6】
? 阳性基因的后续大样本目的区域测序
一项NG发表的研究,科研人员首先对781位银屑病患者以及676位健康对照者的样本进行了外显子测序,获得了众多目标基因。考虑到外显子研究阶段样本量有限,研究者们在第二阶段对9946位银屑病患者和9906位健康对照者的样本进行了目的区域测序研究【7】。
? 变异的功能研究
变异的功能研究,首先是功能预测【8】。
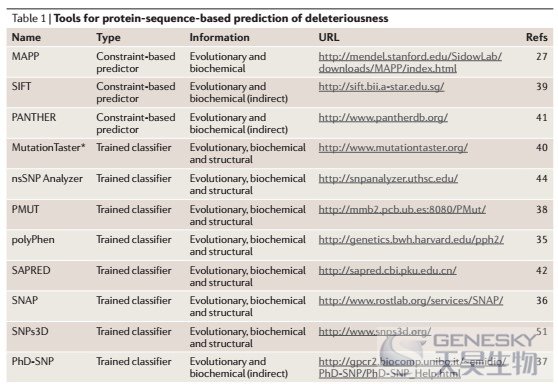
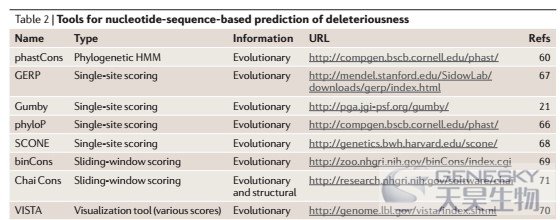
编码区变异功能预测常用工具
序列变异功能预测常用工具
采用功能试验验证变异功能,方法很多,对于不同的复杂疾病,手段也相差很大。后续会单独整理一个帖子。这里先给出两个文献例子让大家参考:
精神类疾病基因突变功能研究:【5】
唇腭裂基因突变功能研究:【9】
关于wes,这里还有个重点要关注!
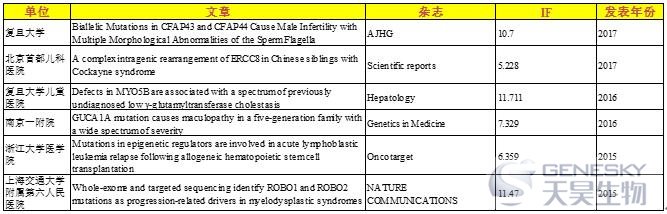
天昊生物在2016年协助客户发表了127篇文章,累计影响因子400+。对于医学基因组测序方向,更是高分文章云集,累计影响因子90+,涉及到了全外显子测序和富有天昊特色的多种目的区域测序技术平台。部分采用天昊wes技术平台的客户文章见下表。
参考文献
【1】Gaynor S C, Breen M E, Monson E T, et al. A targeted sequencing study of glutamatergic candidate genes in suicide attempters with bipolar disorder[J]. American Journal of Medical Genetics Part B Neuropsychiatric Genetics, 2016.
【2】Griswold A J, Dueker N D, Booven D, et al. Targeted massively parallel sequencing of autism spectrum disorder-associated genes in a case control cohort reveals rare loss-of-function risk variants[J]. Molecular Autism, 2014, 6(1):1-11.
【3】Pirooznia M, Wang T, Avramopoulos D, et al. High-throughput sequencing of the synaptome in major depressive disorder[J]. Molecular Psychiatry, 2015, 21(5):650.
【4】Wang L, Li J, Ruan Y, et al. Sequencing ASMT identifies rare mutations in Chinese Han patients with autism.[J]. Plos One, 2012, 8(1):e53727.
【5】Peykov S, Berkel S, Schoen M, et al. Identification and functional characterization of rare SHANK2 variants in schizophrenia.[J]. Molecular Psychiatry, 2015, 20(12):1489-1498.
【6】Deep resequencing of GWAS loci identifies independent rare variants associated with inflammatory bowel disease. Nat. Genet. 43, 1066–1073.
【7】Tang H, Jin X, Li Y, et al. A large-scale screen for coding variants predisposing to psoriasis.[J]. Nature Genetics, 2014, 46(1):45.
【8】Muth T, Benndorf D, Reichl U, et al. Searching for a needle in a stack of needles: challenges in metaproteomics data analysis.[J]. Molecular Biosystems, 2013, 9(4):578-585.
【9】Leslie E J, Taub M A, Liu H, et al. Identification of Functional Variants for Cleft Lip with or without Cleft Palate in or near PAX7, FGFR2, and NOG by Targeted Sequencing of GWAS Loci.[J]. American Journal of Human Genetics, 2015, 96(3):397-411.
【10】Do R, Kathiresan S, Abecasis G R. Exome sequencing and complex disease: practical aspects of rare variant association studies[J]. Human Molecular Genetics, 2012, 21(R1):R1.

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886