摘要:Nature封面:大麦基因组研究最新进展
【昊阅读】Nature封面:大麦基因组研究最新进展,液相捕获芯片有作为
2017年4月27日,Nature以封面文章的形式发表了大麦基因组研究最新成果(A chromosome conformation capture ordered sequence of the barley genome),这是继2012年11月Nature发表大麦参考基因组以来的又一力作。
研究背景
虽然2012年Nature已经发布过大麦基因组图谱,但是由于当时技术的局限,没有很好的对大麦基因组中存在的大量重复元件和着丝粒区域进行测序。本研究采用多种新的测序方法,对上述区域进行了重新测序组装,得到更高质量的大麦参考基因组。
研究材料及平台
研究材料:大麦 barley (Hordeum vulgare L.)
仪器平台:Illumina MiSeq, HiSeq2000 and HiSeq2500 platforms,Roche 454 Titanium,PacBio RS II,Irys platform (BioNano Genomics)
研究内容
1、大麦染色体尺度的基因组组装
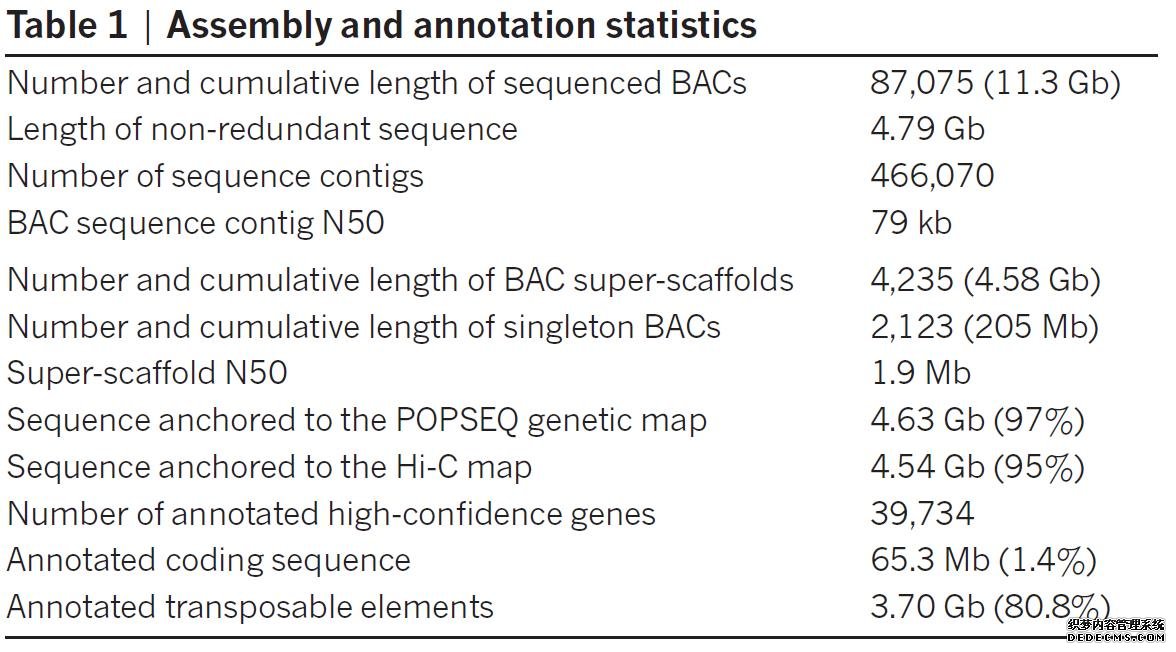
研究人员首先用paired-end和mate-pair测序方法,对87,075个BACs进行测序,之后用群体基因组测序(POPSEQ)技术将scaffold N50从79 kb提高到1.9 Mb,最后利用染色体构象捕获测序(Hi-C)技术在染色体层面完成6,347个super-scaffolds的组装(表1)。
表1、大麦基因组组装及注释统计表
2、大麦基因组重复元件分布情况
许多植物基因组包含了大量拷贝的重复元件,如长末端重复序列(LTR)反转录转座子和DNA转座子,本研究通过对100-150 kb的BACs序列的重新构建,更好的摆脱了大量拷贝数近似的重复元件的影响,成功获得了3.7 Gb的组装序列。图1即为各类重复元件在基因组上的分布情况(图1-a),以及转座元件上下游基因数量分布情况(图1-b)。
图1、大麦基因组重复元件分布情况图
3、应用液相捕获芯片进行大麦品种的分子遗传多样性和单倍型分析
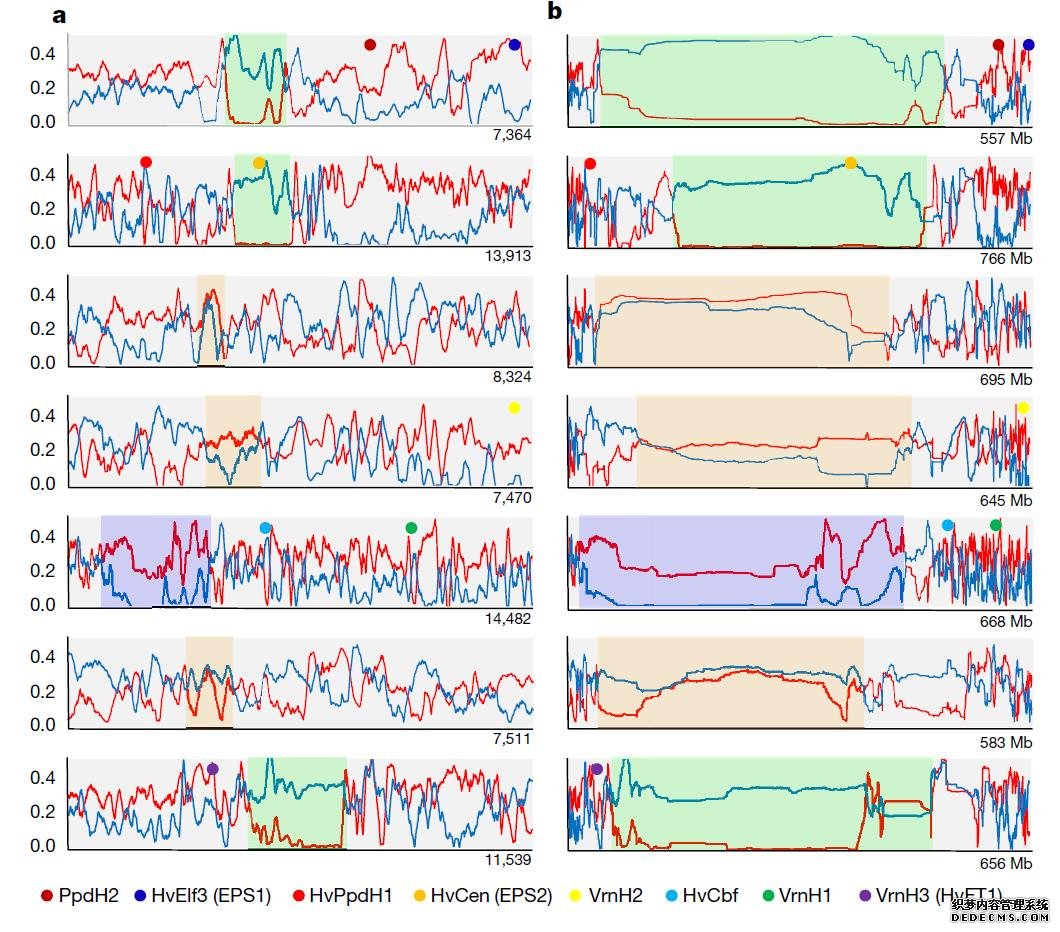
为了探讨新的大麦参考基因组在遗传育种上的应用,研究人员首先通过比较基因组学的方法,扩充了大麦中的基因家族,如重要的抗病基因(NBS-LRR and thionin genes)和硫氧还蛋白基因(thioredoxin genes)家族,之后利用液相捕获芯片对48个冬大麦(图2蓝线)和48个春大麦(图2红线)品种的遗传多样性和单倍型分析。通过对大麦的7条染色体的SNP分型分析,发现了冬大麦和春大麦分子遗传多样性区域分布的异同(图2:分别用绿色、橙色和紫色区域内蓝线和红线表示)。
图2、大麦基因组遗传多样性分布图
研究意义:
大麦作为世界上最重要的粮食作物之一,涉及到人类重大粮食安全问题。大麦基因组图谱的高质量完成,将为科研人员更好的研究大麦功能基因和遗传进化和育种,进而为提高大麦产量、增强大麦抗虫抗病能力、增加大麦营养价值提供坚实的基础。
天昊生物--全外显子组测序和目的区域富集测序的优质服务提供商!
我们通过将全基因组外显子区域DNA,或者个性化定制目标区域DNA进行捕捉富集,在进行文库构建后利用Illumina高通量二代测序系统进行测序,对所获得测序数据进行生物信息学的读取、质量控制及后续遗传学分析。
目前我们可以提供的个性化定制液相捕获芯片服务物种包括:人(H. sapiens)、大鼠(R. norvegicus)、老鼠(M. musculus)、拟南芥(A. thaliana)、牛(B. taurus)、线虫(C. elegans)、旋木雀(C. familiaris)、狨猴(C. jacchus)、果蝇(D. melanogaster)、斑马鱼(D. rerio)、鸡(G. gallus)、恒河猴(M. mulatta)、青鳉(O. latipes)、水稻(O. sativa)、酿酒酵母(S. cerevisiae)、裂殖酵母(S. pombe)等,其他有参物种均可以手工设计。

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886