【科研小助手】二代测序数据质控的那些事
当我们获得二代测序的原始数据之后,除了关心数据量够不够外,测序数据的质量也是我们需要重点关注的,它是我们获得正确结论的基础和保证。那么到底我们需要关注哪些参数指标?怎样的数据可以认为是好的数据呢?今天我们就聊一聊二代测序数据质控的那些事。
目前天昊生物在对llumina测序平台产生的原始数据质控主要采用由英国剑桥Babraham研究所开发的FastQC软件进行,下载网址:http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (图1)。
图1、FastQC软件下载网页
FastQC作为一款设计精巧的数据质量控制软件,可以高效的对二代测序数据的基本信息进行统计,并给出相应的图表及报告。由FastQC处理的数据已经获得Nature、Cell等顶级期刊认可,因此得到了越来越多的生物信息学分析者的青睐。
当我们下载并安装好FastQC后,我们可以对测序数据进行多方面的分析判断,通过的结果用绿色“PASS”表示,不通过的用红色“FAIL”表示,质量位于两者之间的为黄色“WARN”表示。
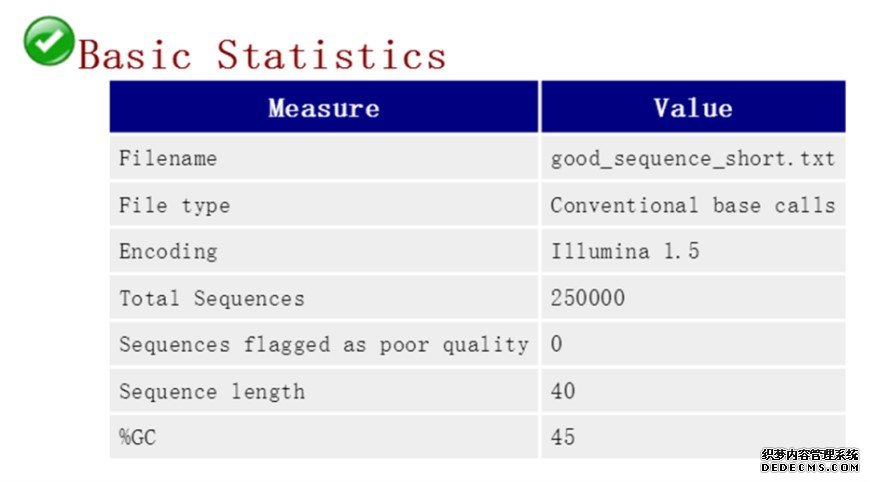
1、原始数据的基本统计信息
包括数据的名称、类型、质量编码格式、reads数目,未通过的reads数目,reads读长及GC含量平均值等(表1)。通过该统计表我们可以对测序的数据质量有一个大致的了解。
表1、基本信息统计表
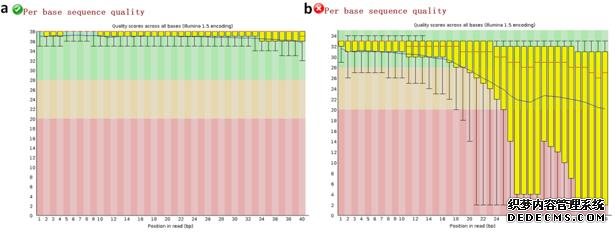
2、原始数据碱基质量分布
Illumina测序平台在获得原始测序序列的同时,会同时给出测序质量的对应信息,具体到每个碱基会给出一个碱基质量值,用Q表示。质量值的计算公式为:Q = -10*log10(E),E为测错的概率,即一条read某位置出错概率为0.01时,其Q值就是20。同理,当出错概率为0.001时,其Q值就是30。
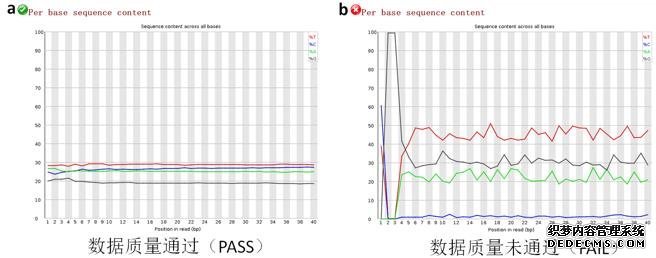
原始数据碱基质量分布图(图2-a,b)的横轴为测序Reads的不同碱基位置,纵轴则显示所有Reads在每一位置的测序质量值统计。因此它反映的就是reads不同碱基位置的质量值。通过图2a与2b的比较,我们就可以直观的了解测序数据质量的好坏。
图2、原始数据碱基质量分布图
该图采用箱线图的方式来展示所有碱基质量的分布,其中黄色箱体的上下两条边框分别代表该位点所有碱基质量的上四分位数和下四分位数,红色的线代表该位点所有碱基的中位数,蓝色的线代表该位点所有碱基质量的均值,黄色箱子上方和下方的黑线分别代表该位点碱基质量的最大值和最小值。背景色根据碱基质量的大小分为绿色、黄色和红色三部分,绿色代表碱基质量在28以上,处于绿色区间说明该位点碱基质量较高;黄色代表碱基质量在20-28之间,处于黄色区间说明该位点碱基质量稍差,但是也属于可接受范围;红色代表碱基质量在0-20之间,此时的碱基质量比较差,序列的可信度不高,可能影响下游分析的准确性,可考虑去除这样的低质量序列。
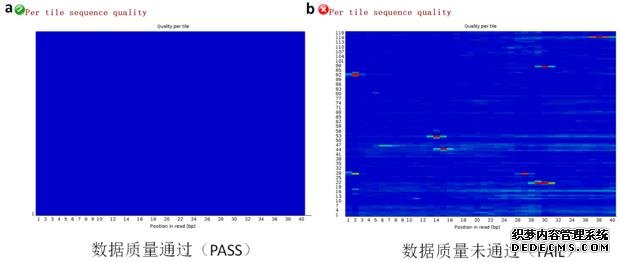
3、原始数据的位置质量分布
图3-a,b是对测序的Flowcell芯片不同小区(tile)位置的测序质量进行评估,显示的是测序的系统误差。一般情况下,商业公司测序芯片有出厂质检,该项均为通过状态,同时,也可以对原始的测序数据进行处理,将测序质量低的Reads进行去除。图3中,蓝色部分是质量较好的点,红色越明显则表示该小区位置测序质量越低。
图3、原始数据位置质量分布图
4、原始数据Reads质量分布
该项统计是对不同测序质量的Reads进行了分布统计(图4-a,b)。横轴显示的是每条Read平均的碱基质量值,纵轴则是在该质量下的reads数目分布。因此,Read数据质量越高,其红线的主要部分越向右偏移。反之则Read数据质量越低,其红线表示的主要部分越向左偏移。
图4、原始数据Reads质量分布图
5、Reads相同位置碱基比例
图5-a,b是对测序的ATGC四种碱基在Read每个位置的比例进行统计。横轴是Reads不同的测序碱基位置,纵轴是该位点的所有Reads的四种碱基比例情况,不同颜色代表不同的碱基类型。理论上A和T碱基相同,G和C含量相同。同时,整体含量应该与整个基因组水平类似,且相对稳定不变,呈水平线。但由于与测序的引物接头相连,因此可能会有些波动,属于正常波动,但如果整体范围内剧烈的波动,并且某种碱基比例明显偏离,则说明质量较差。
图5、Reads相同位置碱基比例图
6、Reads平均GC含量分布图
该项指标是对所有Reads的平均GC含量分布情况进行的统计(图6-a,b)。横轴是平均GC含量百分比,纵轴是该GC含量的Reads数目。理想的情况是测序的实际结果(红线),应与理论结果(蓝线)相似, 质量较差的结果则表现为两条线的相似度不好。
图6、Reads平均GC含量分布
7、Reads中N含量分布
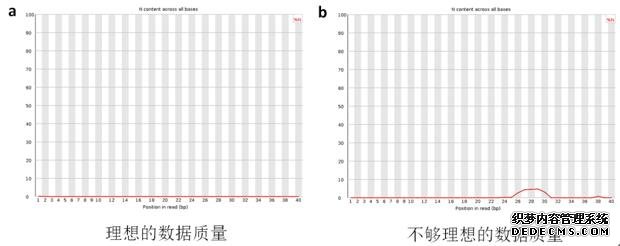
N代表的是测序无法辨别的碱基,该项检测是对所有Reads的N含量百分比进行统计,横轴是Reads中碱基的位置,纵轴是N的比例。理想的测序结果,红线的纵轴指示几乎为零(图7a)。并且测序Reads的长度为自己的目标长度, 不够理想的结果是红线有较大起伏波动(图7b)。
图7、Reads中N含量分布图
8、Reads的长度分布
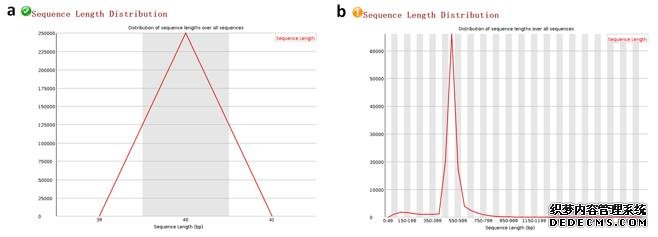
该项指标较容易理解,即所测Reads长度的分布统计(图8)。它能较好的反应测序整体长度,该项指标通常在短Reads测定中比较理想,而在罗氏454等测序仪中,因为测序片段较长,需要更加关注。
图8、Reads的测序长度分布图
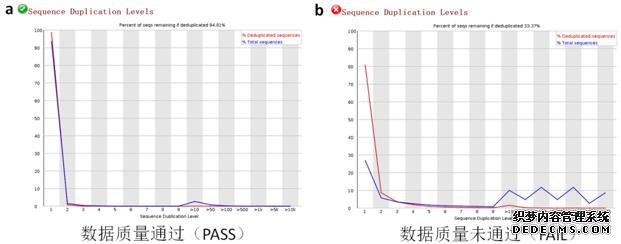
9、序列重复水平分布及overrepresented序列统计
序列重复水平分布是统计序列完全一样的Reads的频率,通过该指标可以提示我们测序过程产生的偏差,如在建库PCR过程中可能产生的偏差等(图9,表2)。横轴是序列的重复水平,数字代表重复次数,大于10次时按不同代表次数合并展示。纵轴是该重复水平下的Reads数目百分比(红线表示所有序列, 蓝线表示去重复后序列),这里的Reads通常只取fq数据的前100,000条代表性的Reads进行统计。理想的数据质量下,测得的Reads的重复次数应该很少,但当出现随着重复水平增加而测得的代表性Reads比例又较高时,则表明可能数据不够理想。当然这里排除特殊建库方法如ChIP-Seq等因素。
图9、序列重复水平分布图
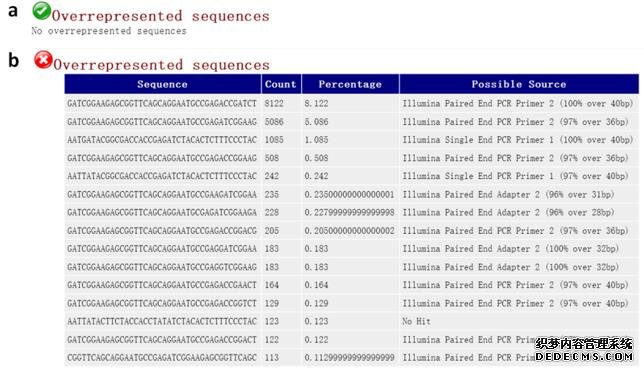
Overrepresented序列统计表显示的是同一序列Reads数占总测序Reads数大于0.1%的统计表,它则主要列举了可能的建库污染相关序列信息。
表2、Overrepresented序列统计表
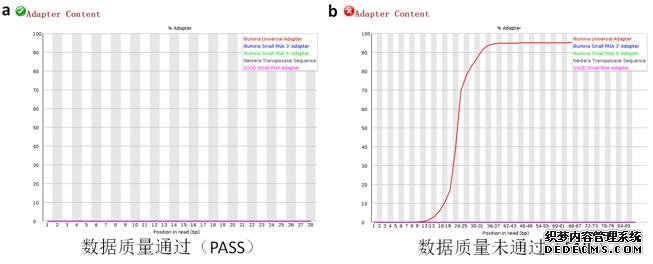
10、测序序列的接头含量分布
图10、测序序列的接头含量分布图
该项指标是对测序序列的接头含量进行统计,旨在检查测序接头是否去除干净。因为测序文库的构建需要有接头的连接,而接头是否去除干净将会对后续的分析产生影响。正常情况下接头的含量应该接近于零(图10-a),如果含量有较大变化甚至陡然增高,测预示着接头未除干净(图10-b)。
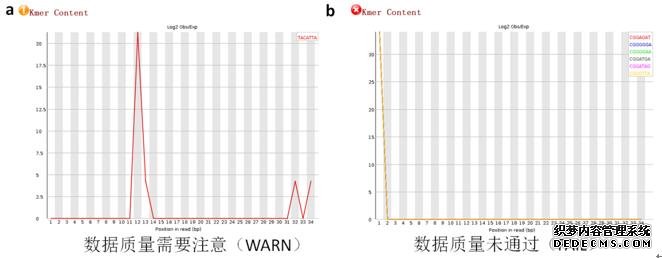
11、Kmer含量分布
11、Kmer含量分布图
该指标也是衡量overrepresented序列的一种方法,它通过检测Kmer在各位置上的数量,并通过二项分布检验来寻找均匀覆盖度情况下的显著偏差。如果某种短序列在Reads中大量出现,并且其频率高于统计期望的话,FastQC将其记为overrepresented Kmer。检测出的短序列越少则数据越好,对于较差质量的数据,软件会给出相应序列列表,并将在分布图的右上角列出最显著的6个偏差序列。
图表数据来源:http://www.bioinformatics.babraham.ac.uk/projects/fastqc/good_sequence_short_fastqc.html#M1
天昊生物一直以来重视对测序数据的质量控制,在前期对样品质检合格并安排上机测序后,通常情况下对获得的二代测序的数据要求为Q30数据达到80%以上,Q20比例达到90%以上,以保证客户对数据质量的要求。

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886