SSR分子标记在遗传多样性研究中的应用
生物多样性的研究和保护是目前全世界普遍关注的问题,其中研究较多的是遗传多样性、 物种多样性及生态系统多样性。 其中遗传多样性是后者的物质基础,生态系统多样性离不开物种的多样性 ,也就离不开不同物种所具有的遗传多样性。微卫星标记具有分布广泛、多态性丰富、呈共显性和检测方便快速等特点,广泛应用于估测品种间遗传距离、构建系统发生树及度量品种遗传多样性。本期小编就介绍一下SSR分子标记技术及其在动植物遗传多样性研究中的具体应用。
1. SSR简介:
简单重复序列,也称微卫星(SSR),其串联重复的核心序列为1-6bp,其中最常见的是双核苷酸重复,即(CA)
n和(TG)
n,在植物基因组中(AT)n最多,长度一般在100bp以内。SSR的产生是在DNA复制或修复过程中DNA滑动、错配或者有丝分裂、减数分裂期姐妹染色单体不均等交换的结果。不同遗传材料重复次数的可变性,导致SSR长度的高度变异性,从而产生了很多等位基因,导致了微卫星的高度多态性。
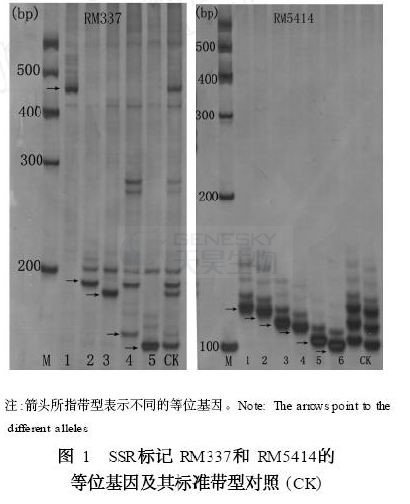
图1 SSR标记RM337和RM5414的等位基因,箭头所指带型代表不同的等位基因
2. SSR标记的基本原理:
尽管微卫星DNA分布于整个基因组的不同位置,但其两端序列多是保守单拷贝序列,因此可以根据这两端的序列设计以对特异引物,通过PCR技术将其间的核心微卫星DNA序列扩增出来,利用电泳分析技术就可以获得其长度多态性。SSR标记的高度多态性主要来源与串联数目的不同。根据分离片段的大小决定基因型,并计算等位基因发生频率。
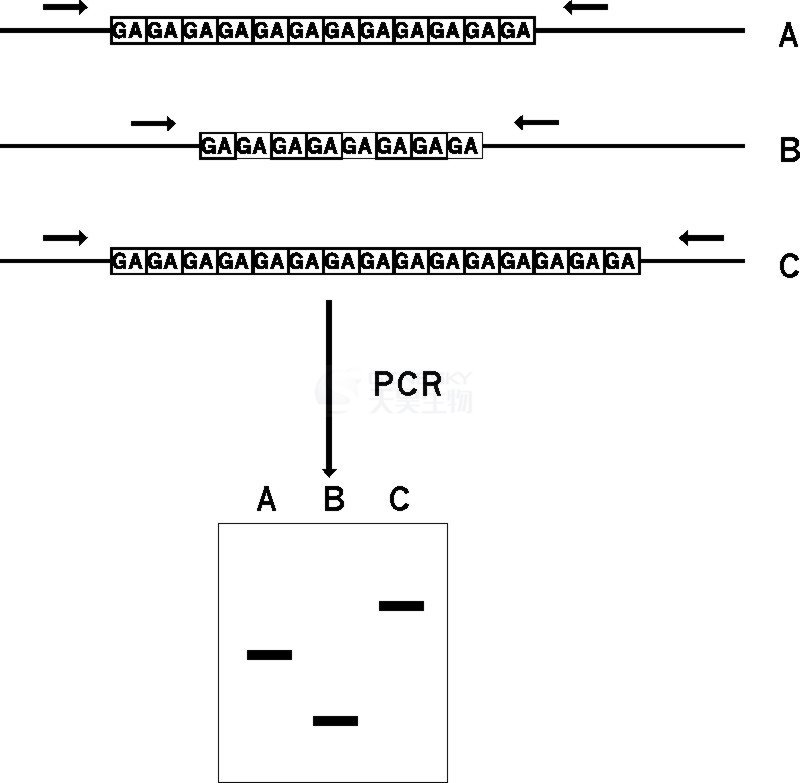
图2 SSR多态分析示意图
3. SSR的分类: 根据SSR核心序列排列方式的不同,可分为3种类型:
1)完全型,指核心序列以不间断的重复方式首尾相连构成的DNA。如:ATATATATATATATATATATATATATATATATAT;
2)不完全型,指在SSR的核心序列之间有3个以下的非重复碱基,但两端的连续重复核心序列重复数大于3 。如:ATATATATGGATATATATATCGATATATATATATATATGGATATATATAT;
3)复合型,指2个或2个以上的串联核心序列由3个或3个以上的连续的非重复碱基分隔开,但这种连续性的核心序列重复数不少于5 。如:ATATATATATATATGGGATATATATATATA 。3种类型中完全型是SSR标记中应用较多的一种类型。
4. SSR标记特点:
l 检测到的一般是一个单一的多等位基因位点。
l 共显性遗传,符合孟德尔遗传规律,所以可以用来鉴别杂合子和纯合子。
l 数量丰富,覆盖整个基因组,而且分布均匀。
l SSR序列的两侧序列常较保守,在同种而不同遗传型间相同。
l 多数SSR无功能作用,增加或减少几个重复序列频率高,因而在品种间具有广泛的位点变异。
5. SSR在植物基因组中的分布:
在植物中,平均23.3kb就有一个SSR;双子叶植物中的SSR数量大于单子叶植物,前者两个SSR之间的平均间距为21.2kb,后者为64.6kb;绝大多数单碱基重复型及2碱基重复型SSR存在于非编码区,3碱基重复型多位于编码区。
6. SSR引物的来源:
1) 首先要确定研究的物种是否有可供参考的SSR序列,有很多物种是已经有文献发表的SSR引物序列,因此不用自己设计引物,尽量选择文献报道比较多的多态性好的位点。
2)如果所研究的物种没有文献报道,这样的话需要自己开发SSR引物:要么借鉴其它近缘种引物序列,要么从该物种基因组序列中利用SSRIT或SSR-Hunter软件筛选SSR位点,要么通过筛选文库、测序开发自己的SSR引物。通过实验验证,最终筛出的位点需要符合如下要求:不连锁、多态性好、易扩增。
7. SSR分子标记检测方法
|
|
高浓度琼脂糖电泳 |
聚丙烯凝胶电泳 |
荧光毛细管电泳 |
SSR-seq |
|
优点 |
1. 便宜
2. 方便
3. 分辨率100bp |
1. 分辨率1-10bp
2. 位点较少时较荧光毛细管电泳便宜 |
1. 单碱基检测
2. 可以适当多重PCR |
1. 单管最多可以做到20多个位点多重PCR
2. 可以看到序列,准确检测位点的具体重复次数
3. 可以找到真正的多态位点,另外可以避免插入缺失引起的序列长度变化误判
4. 2倍体和多倍体物种都可以准确计算每种基因型比例 |
|
缺点 |
1. 分辨率低
2. 具体重复数不知
3. 多倍体物种每种基因型比例不能准确定量
4. 需要把PCR产物一代测序进行验证 |
1. 有毒,配制困难
2. 电泳时间久
3. 单次检测通量低
4. 多倍体物种每种基因型比例不能准确定量
5. 需要把PCR产物一代测序进行验证 |
1. 价格贵
2. 只能看长度,不能看到序列
3. 多倍体物种每种基因型比例不能准确定量
4. 需要把PCR产物一代测序进行验证 |
|
8. 数据分析
根据各SSR标记在相同分子量片段的有无,统计得到所有位点的二元数据,有DNA扩增带记为1,无带记为0,将各材料的SSR条带,转换成“0,1”矩阵后,采用POPGENE软件计算出平均等位基因数(
Na)、有效等位基因数(
Ne)、期望杂合度(
He)、观测杂合度(
Ho)、多态位点数(
AP)、多态位点百分率(
P)、Nei’s基因多样性指数(
H)、Shannon′s信息指数(
I)、核苷酸多样性(
Pi)、遗传分化系数(
Fst)、基因流(
Nm)。采用软件NTSYS软件计算样品间的遗传相似性系数与遗传距离,根据样本间遗传相似系数或遗传距离的大小,可将满足一定条件的样本化归为一个遗传谱系,如相似性大于90%或遗传距离小于10%;采用软件SAHN clustering进行不加权成对算术平均法聚类分析(UPGMA),根据聚类结果分析各样品间亲缘关系及起源。
l 平均等位基因数(
Na):一般来说, 平均每个座位的等位基因数越多,其群体多态性就越丰富 。
l 有效等位基因数(
Ne):是反映群体遗传变异大小的一个指标,其数值越接近所检测到的等位基因的绝对数,表明等位基因在群体中分布越均匀,两者差异大说明等位基因在群体中分布不均匀。
l 观测杂合度(
Ho):指随机抽取的两个样本的
等位基因不相同的概率,观测杂合度(Ho)=观察得到杂合个体数/样本个体数总数。
l 期望杂合度(
He):指理论计算得出的杂合度,He值的范围从0(说明无多态性)到1(说明无限多个等位形式具有相同的频率,是个极限值)。常用期望杂合度来衡量群体的遗传多样性的高低。期望杂合度值越高,反映群体的遗传一致性就越低,其遗传多样性就越丰富。
l 多态信息含量(
PIC):是指微卫星DNA变异程度高低的一个指标,反映微卫星DNA多态高低。当某微卫星座位PIC >0. 5时,即表明该座位为高度多态座位;0. 25 < PIC < 0. 5时 ,为中度多态座位;PIC < 0. 25时,该座位为低度多态座位。
l Nei’s基因多样性指数(
H):Nei是指通过计算遗传距来分析遗传多样性的,即通过计算单倍型多样性指数来计算群体间的核苷酸序列歧化距离,是根据种群间不同基因所占的比例算出的遗传多样性。
l Shannon′s信息指数(
I):群体遗传多态度用Shannon′s 多样性指数表示,Shannon′s 多样性指数= - ∑Xi ln ( Xi / n) ,式中X i 为位点i 在某一群体中出现的频率,n 为该群体检测到的位点总数。
l 核苷酸多样性(
Pi):任意两序列间的平均核苷酸差异数,称为核苷酸多样性。是衡量群体内核苷酸多态性水平高低的重要指标。
l 多态位点数(
AP):某个位点有两个以上
等位基因,该位点就称为多态性位点
l 多态位点百分率(
P):多态性位点占总位点数的百分比即为多态性位点百分率,是反映遗传多态性的重要指标。
l 遗传分化系系数(
Fst):代表不同群体间的遗传分化水平,Fst的值是0-1,Fst越小,亚群间分化程度越高,存在着较小的基因流水平。
l 基因流(
Nm):指生物个体从其发生地分散出去而导致不同种群之间基因交流的过程,可发生在同种或不同种的生物种群之间。基因流的基本作用是消弱了种群间的遗传差异。岛屿模型中的Nm为每代迁入的有效个体数,即基因流的估计值,Nm=1/4(1/Fst-1)。
9. SSR标记的应用
目前SSR分子标记已成为系谱分析、分类鉴定、亲缘关系分析、体细胞杂种鉴定、遗传图谱构建、基因定位、育种材料早期选择首选的分子标记之一。SSR技术已在苹果、梨、桃、杏、葡萄、柑橘、称猴桃、板栗、核桃、樱桃、山植、椰子等果树上有了很多的应用。
遗传多样性研究
利用分子标记,可以从群体的遗传变异角度,从分子水平上研究物种的遗传多样性,探讨种内及其近缘种属植物的起源与进化。刘勇[1]等利用SSR分子标记对63份江西柑橘地方品种和野生近缘种的遗传多样性进行了研究,采用30对引物共检测到374条等位基因,采用UPGMA进行聚类,将份供试材料分为8个不同类群。随后又采用31对SSR引物对122份柏类种质资源及其近缘种的遗传多样性进行了分析研究,共检测到335个等位基因变异,平均每个位点检测到9.85个等位基因。
核心种质建立
分子标记技术可被用于群体核心种质资源的构建,构建后的核心种质资源要求以最少的数量可以最大限度地代表供试资源的遗传多样性。李银霞[2]等采用分子标记技术,构建了桃品种核心种质,对56份桃初级核心种质的形态农艺性状数据和等位基因进行了分析,研究了采用不同聚类方法和完全随机取样方法下种样本群体的遗传多样性指数、保留比例和各频率段等位基因丢失的比率。
系谱分析
果树资源中有大量的天然杂种,在实际生产中,有时对一些品种的亲本无法加以判断,因此可利用品种的指纹图谱,对其进行相应的聚类分析,可以对供试材料进行聚类分析,以确定物种之间的亲缘关系。俞明亮[3]等用SSR分子标记技术对供试桃种间的亲缘关系进行了的分析研究,指出桃亚属间普通桃与新疆桃之间亲缘关系最近,陕甘山桃与甘肃桃之间也有较近的亲缘关系。
品种鉴别
果树属于无性繁殖植物,新老品种数量繁杂众多,极易造成同物异名或同名异物的混乱现象,合理的利用分子标记技术可以有效的鉴别果树品种系。Botta[4]等利用4对SSR引物对意大利的24个葡萄品种进行了鉴定分析,结果表明其中3个品种的扩增片断一样,据此判定三者可能属于同物异名,其它品种均能被区分开来。
参考文献:
[1] 刘勇,吴波,刘德春,等江西柑橘地方品种资源及野生近缘种SSR分子标记.江西农业大学学报,2005,27(4):486-490.
[2]李银霞,安丽君,姜全,等.桃品种核心种质的构建与评价.中国农业大学学报,2007,12(5):22-28.
[3]俞明亮桃性状遗传评价和分子标记技术研究.江苏南京农业大学,2004
[4]Botta R, Scott NS,et al.Evaluation of microsatellite sequence-tagged site markers for characterizing Vitis vinifera cultivars. Vitis,1995,34(2):99-102.
天昊生物SSR分型部分发表文章:
1. Feedback activation of leukemia inhibitory factor receptor limits response to histone deacetylase inhibitors in breast cancer. Cancer Cell, 2016 Sep 12;30(3):459-73. doi: 10.1016/j.ccell.2016.08.001.(IF= 23.214 )
2. c-Myc alteration determines the therapeutic response to FGFR inhibitors.Clinical Cancer Research. 2016 Jul 11. doi: 10.1158/1078-0432.CCR-15-2448. (IF= 8.738)
3. JX06 selectively inhibits pyruvate dehydrogenase kinase PDK1 by a covalent cysteine modification. Cancer research. 2015 Nov 15;75(22):4923-36. doi: 10.1158/0008-5472.CAN-15-1023. (IF= 8.556)
4. Triptolide Induces Cell Killing in Multidrug-Resistant Tumor Cells via CDK7/RPB1 Rather than XPB or p44. Molecular cancer therapeutics, 2016 Jul;15(7):1495-503. doi: 10.1158/1535-7163.MCT-15-0753. (IF= 5.58)

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886