
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886

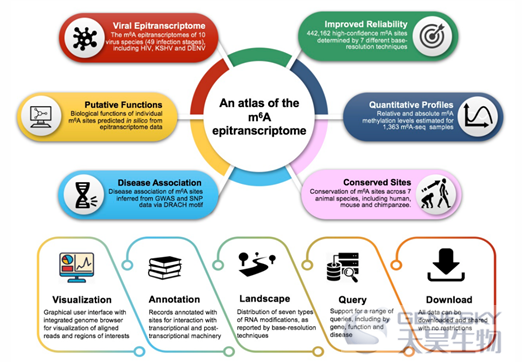


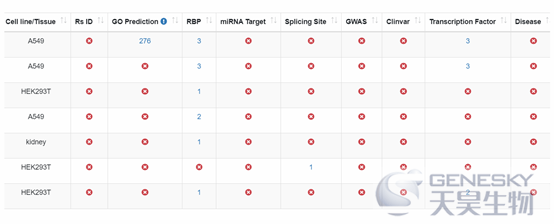
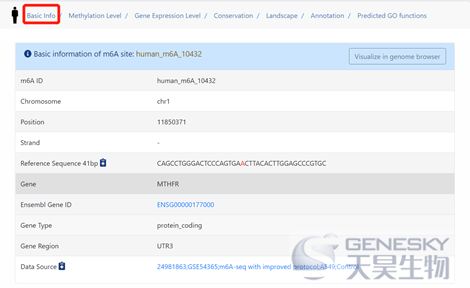
点击某个m6A位点查看细节:
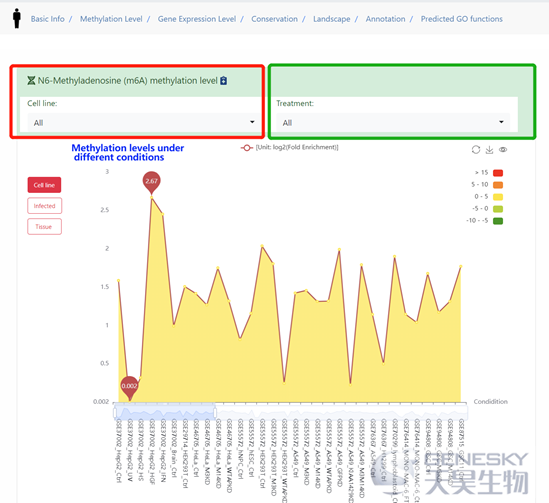
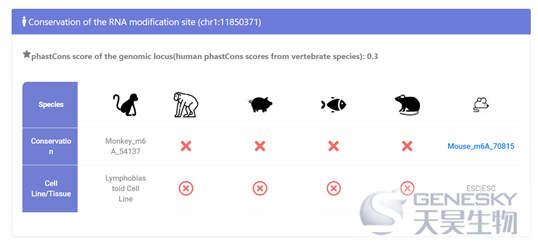
d:位点保守性情况
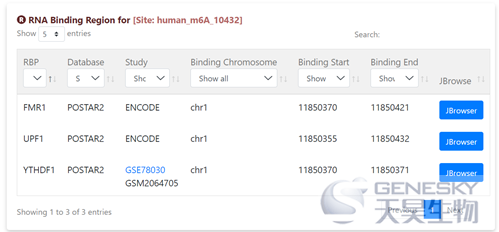
e:位点附近的其他RNA修饰情况和转录后调控信息

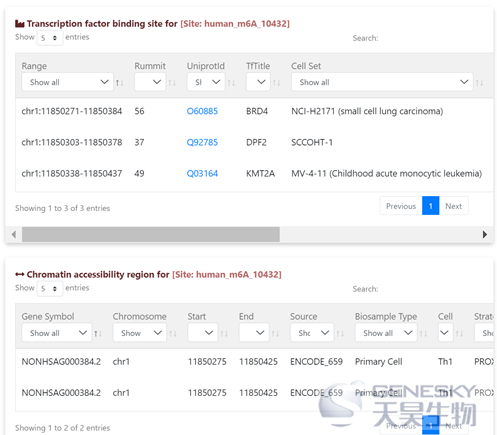
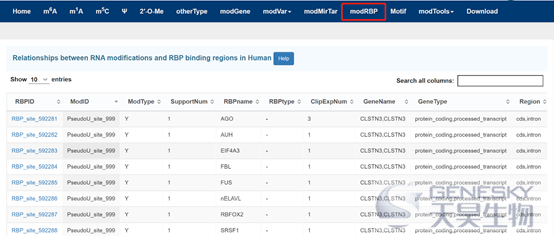
f:位点注释信息(RBP信息,转录因子结合信息,染色质可接近信息等)
g:GO功能信息

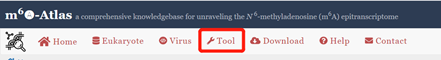
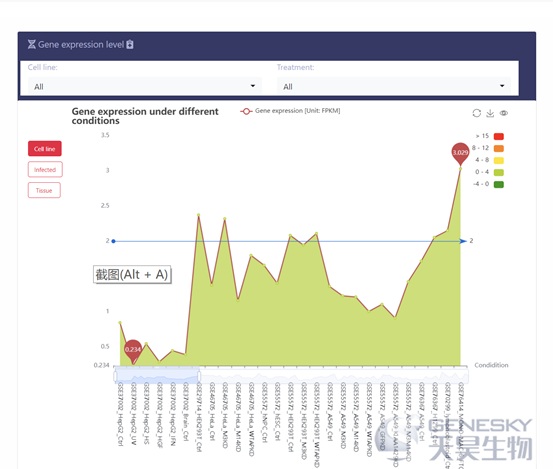
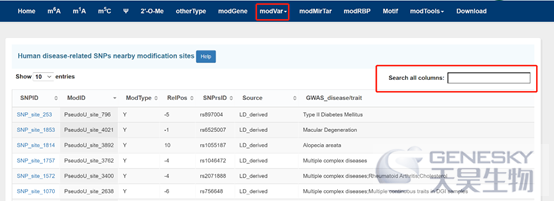
(1)可以用来分析自己数据中的m6A位点保守性情况
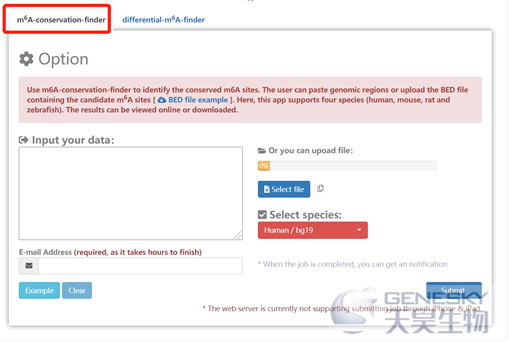
(2)在线进行差异m6A图谱分析
● 网址:http://rna.sysu.edu.cn/rmbase/
● 开发团队:中山大学生命科学学院屈良鹄教授团队
● 参考文献:RMBase v2.0: Deciphering the Map of RNA Modifications from Epitranscriptome Sequencing Data. Jia-Jia Xuan, Wen-Ju Sun, Ke-Ren Zhou, Shun Liu, Peng-Hui Lin, Ling-Ling Zheng, Liang-Hu Qu*, Jian-Hua Yang*.
● 数据库功能:RMBase v2.0整合了大量的表观转录组测序数据,探索RNA的转录后修饰,以及它们与microRNA结合、疾病相关SNPs和RNA结合蛋白(RBP)的关系。RMBase v2.0扩展了566个数据集,包括来自47项研究13个物种的1 397 244个修饰位点,比上一版本扩展了约10倍。它包含约1 373 000个N6-甲基腺苷(m6A)修饰位点信息、5 400个N1-甲基腺苷(m1A) 修饰位点信息、9 600个假尿苷(Ψ) 修饰位点信息、1 000个5-甲基胞嘧啶(m5C) 修饰位点信息、5 100个2′-O-甲基化(2′-O-Me)和约2 800个其他100种类型的修饰。
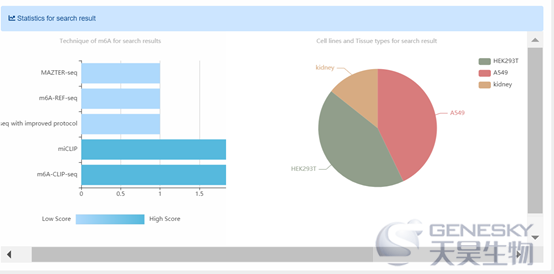
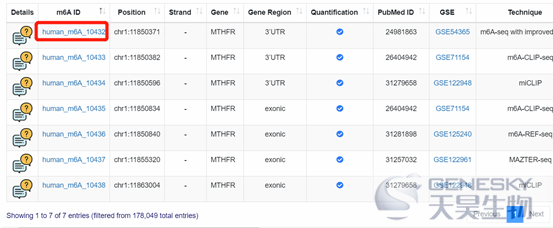
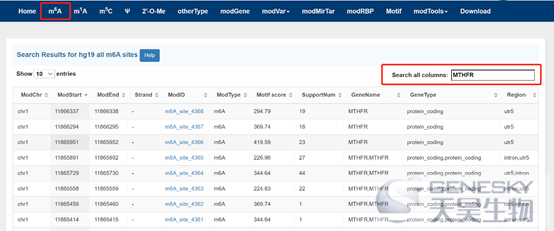
搜索m6A位点:列表形式搜索


创新基因科技,成就科学梦想
微信扫一扫
关注该公众号