SSR和SNP检测要两手抓,两手都要硬!--文章解读:SSR和SNP分子标记在真菌群体遗传结构中的作用比较
摘要: SSR和SNP分子标记比较
今天小编给大家分享一篇2017年8月16在Heredity(IF=3.961)上发表的文章,研究人员通过比较SSR和SNP两种分子标记在真菌群体遗传多样性分析中的结果,发现
SSR和SNP这两种分子标记在种群遗传结构研究中各有优势!在您的研究中,或许
SSR和SNP检测也要两手抓,两手都要硬!
研究背景: 在过去的几年中,简单重复序列(SSR,也被称为微卫星)和单核苷酸多态性(SNPs)已成为用于研究生物群体中的遗传变异最常用的分子标记。然而,只有少量研究比较了这两种分子标记在野生种群遗传结构中的不同,而且没有针对真菌研究的相关报道。
科学问题: (1)本实验开发的两组分子标记是否足以描绘种群遗传结构?(2)SNP和SSR标记在种群的遗传分化和遗传结构方面是否能够得到相似结果?(3)如果存在差异,产生的原因可能是什么?
实验目的:本文研究人员评估了SSR和SNP对蜜环菌(
Armillaria cepistipes)在不同的空间尺度种群遗传结构中的作用。
实验材料: 研究人员分别在乌克兰喀尔巴阡山脉较小区域(150 km
2)和瑞士阿尔卑斯山地区较大区域(41000 km
2)共收集407个样本(图1)。
图1、采样地点(左下图显示的是阿尔卑斯山地区种群取样情况。白色点表示北方亚群(North),绿色点表示南方亚群(South)。右下图显示的是喀尔巴阡山脉地区种群取样情况。蓝色点表示山毛榉亚群(Beech),橙色点表示混合/针叶树亚群(Mixed/conifer))
实验结果:
1、SSR和SNP分子标记筛选
从24个单拷贝蛋白编码基因中选择131个SNP位点,进行全部样本的检测。结果在78%样本中成功得到117个位点(89%)的扩增结果。其中,31个用于后期基因分型检测,其中在所有样本及之后的32个样本重复中得到97%成功率,等位基因读取差别<1%,所有31个SNP位点是二等位,其中29个的最小等位基因频率(MAF)在5%以上。
根据之前的文章报道,从基因组草图中发现281个SSR位点,筛选出17个多态性SSR位点用于后续基因分型,其最常见等位基因多态性频率低于95%,
2、遗传多样性分析
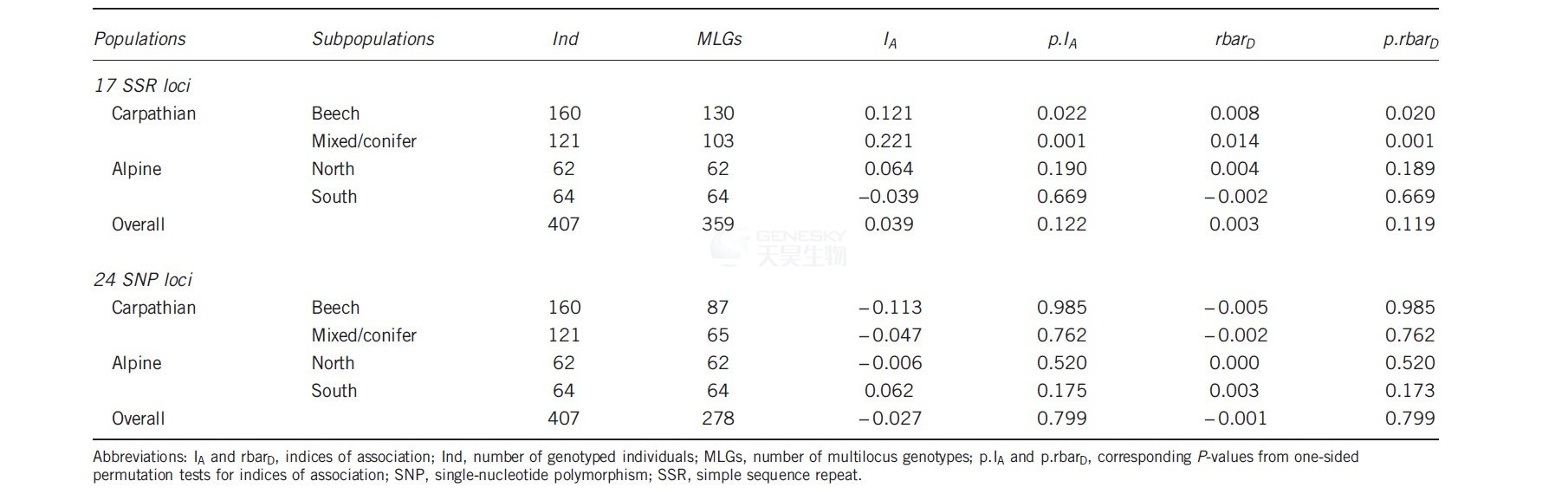
用17个SSR对407个样品进行的基因分型发现,359个样品显示出等位基因的特殊组合,而只有278个样品存在SNP多等位基因型区别(表1)。
表1、蜜环菌不同种群SSR和SNP标记多位点基因型汇总表
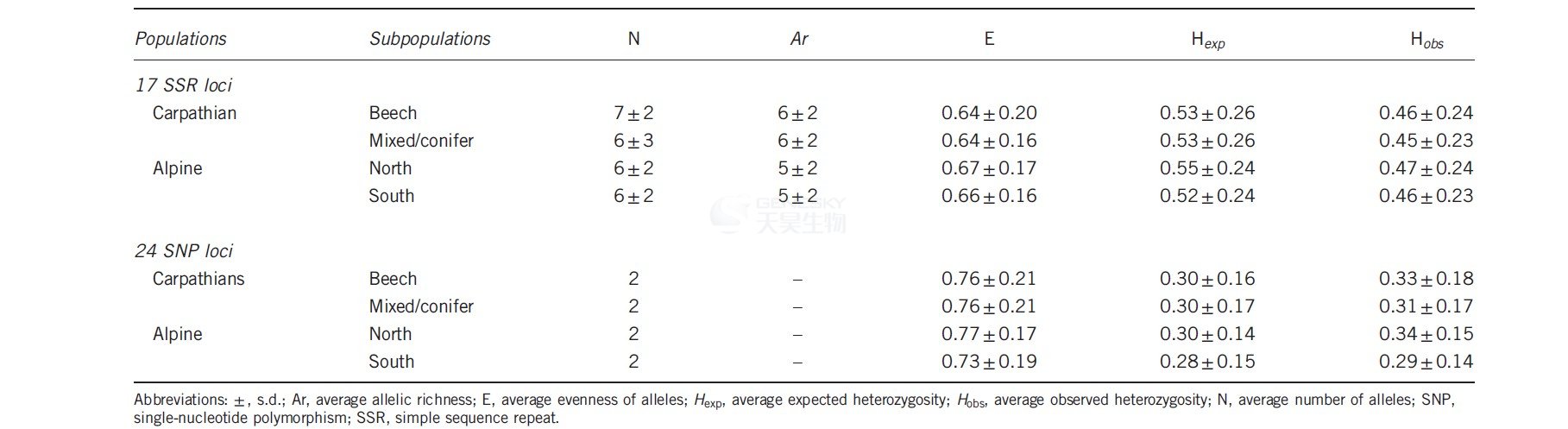
4个亚群SSR位点的平均等位基因丰度(
Ar)相似(表2),但是SSR之间的变化有所不同。SSR和SNP在等位基因平均均匀度(E)和平均期望杂合度(
Hexp)变化很小,但位点之间变化很大。在亚群之间,SSR位点显示出比SNP位点更高的平均观测杂合度(
Hobs)。
表2、蜜环菌不同种群SSR和SNP标记情况表
3、群体遗传结构分析
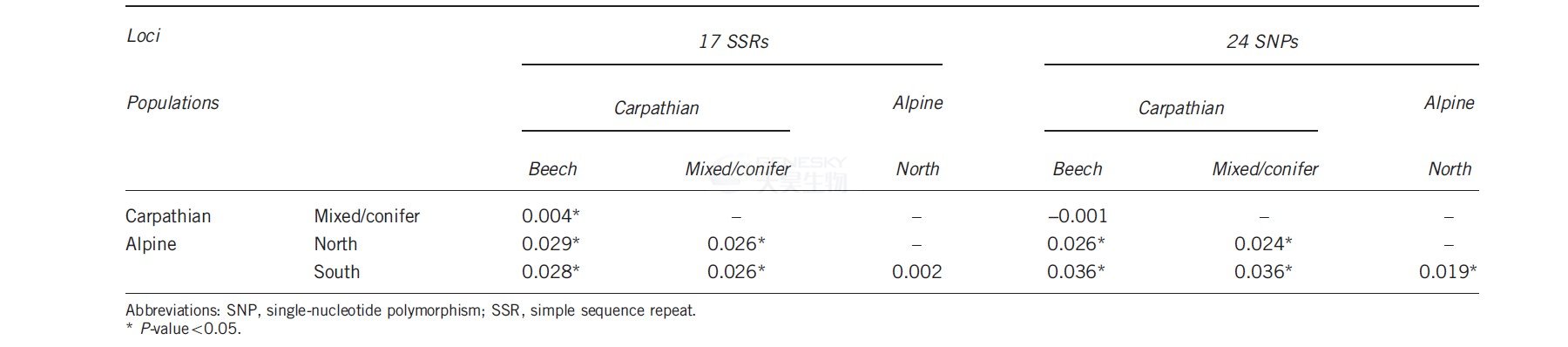
两种分子标记在亚群间的
FST都较低,即便在两个地理位置差别大的种群中也是如此(表3)。
表3、蜜环菌不同亚群SSR和SNP标记遗传分化系数FST对比表
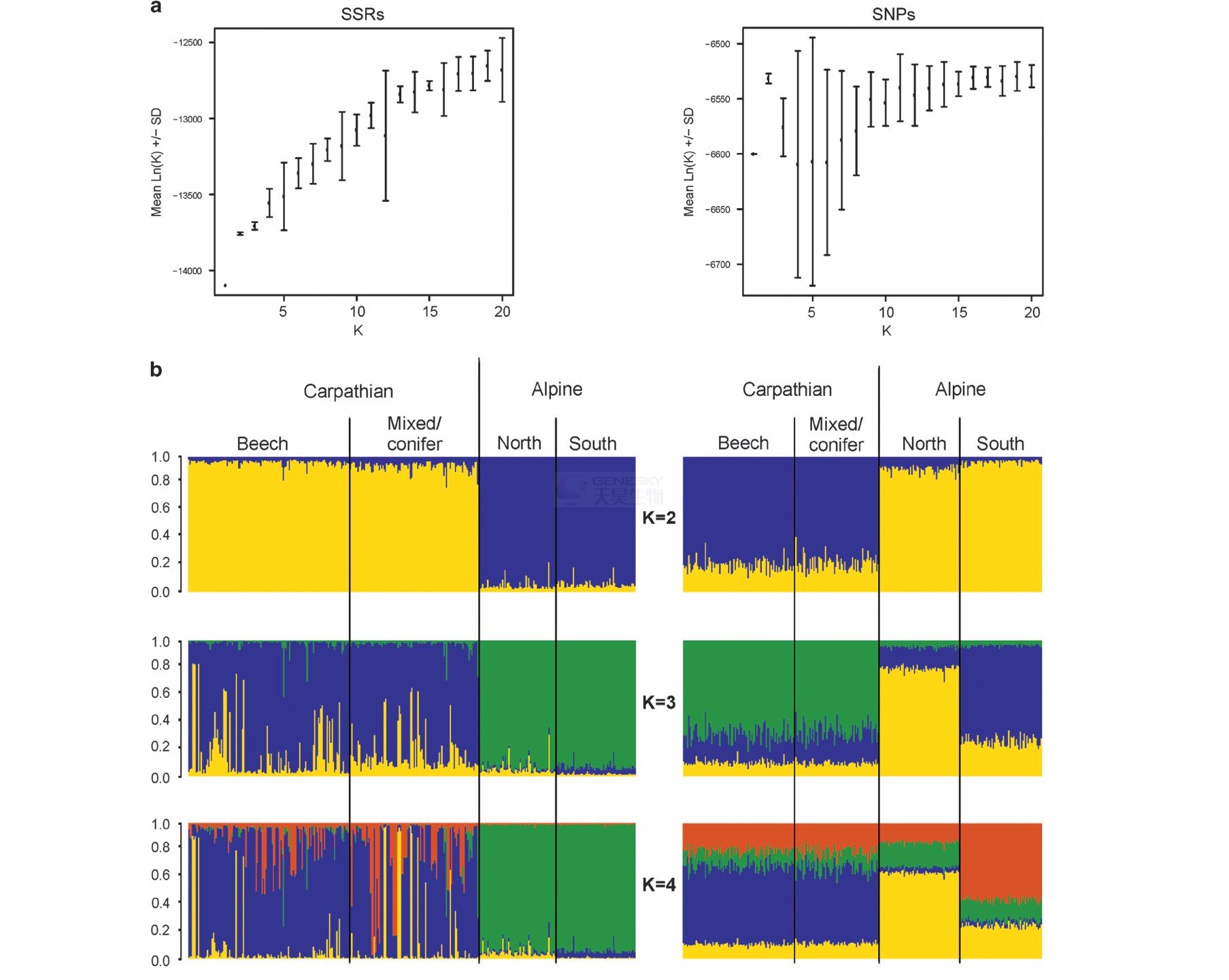
通过对所有4个亚群的遗传结构分析结果后发现,用SSRs分析的基因簇比SNPs的要多(图2)。SSR结果显示其对数似然比稳定的上升到20个基因簇,但其间几个s.d值较大。而SNP结果在K>3时对数似然比不够稳定。遗传结构结果显示,当K=4时,SSR能够将359MLGs较好的根据地理位置区分开,而当K=3时,基于SNP数据可以将两个地区种群清晰的分开(图2b)。
图2、蜜环菌不同亚群SSR和SNP标记STRUCTURE分析图
a) 不同基因簇个数平均似然对数散点图。b) 个体归属特定基簇平均估算成员概率柱状图
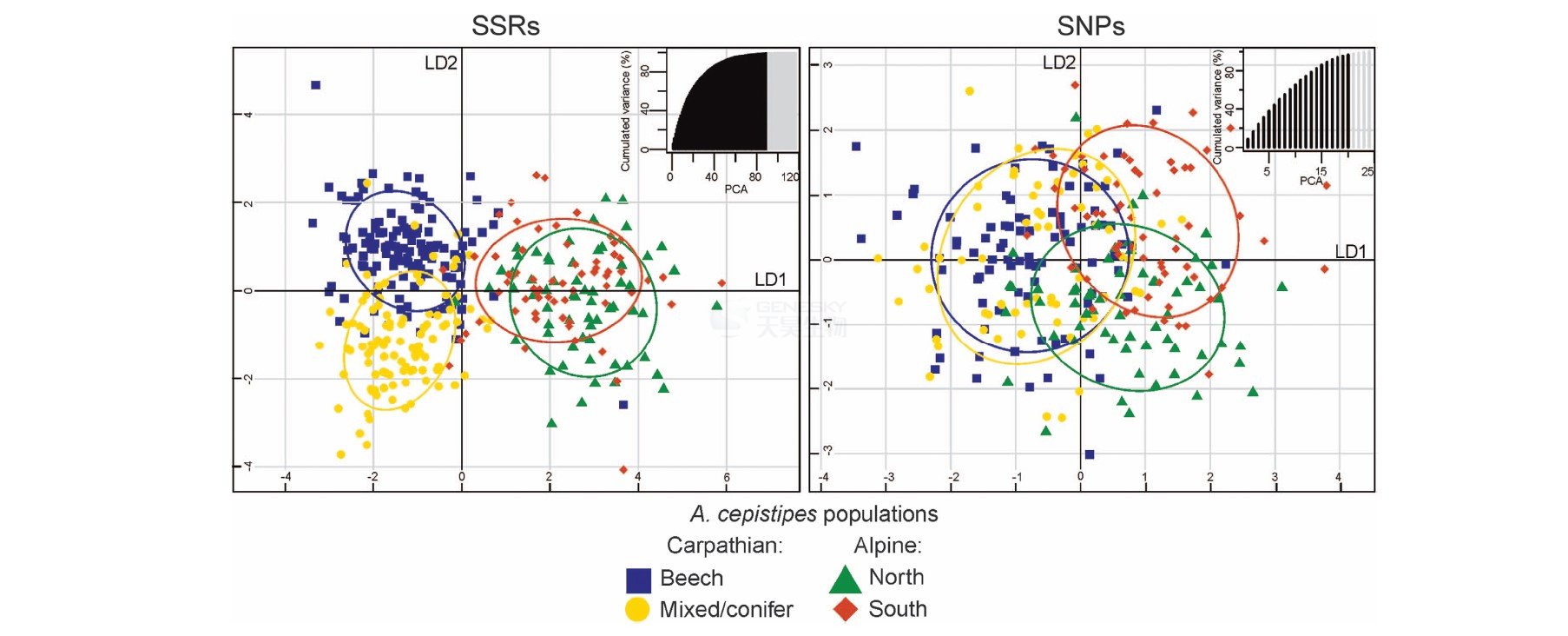
两种分子标记的主成分判别分析表明,基于SSR数据的喀尔巴阡山脉地区两个亚群在第二个轴上可以更好区分,而阿尔卑斯山地区重叠的更多。相反,基于SNP数据结果表明,喀尔巴阡山脉地区两个亚群基本重叠,而阿尔卑斯山地区区分的更好(图3)。表明SSR标记似乎更适合检测小空间尺度的真菌种群遗传结构,而SNP标记在检测更适合大尺度范围的遗传结构分析。
图3、蜜环菌不同亚群SSR和SNP标记主成分判别分析图
主散点图代表前两位主成分的线性判别个体分布,右上角为主成分交叉验证判别分析图
文章总结:
对获得的菌株分别用基因组中的17个SSR位点和不同单拷贝保守基因中的24个SNP位点进行分析,这两种标记在不同空间尺度中表现出不同的遗传结构模式,
多等位基因SSR标记似乎更适合检测小空间尺度的真菌种群遗传结构(区域半径约50 - 100 km),而SNP标记在检测大尺度范围(区域半径约1000 km)中的真菌种群遗传结构更具优势。尽管存在这些差异,这两种标记类型均适用于检测两种种群遗传结构较弱的蜜环菌。
关于天昊:
上海天昊生物拥有基于一代和二代测序平台的SSR和SNP检测能力,为不同样本量和不同位点数量提供有针对性解决方案,期待着与您的合作。

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886