TCGA数据库弄不临清,怎么好意思在肿瘤圈social?---上篇
如今,肿瘤研究者不知道癌症基因组图谱集(The Cancer Genome Atlas,TCGA)数据,就如同时尚达人不知道YSL口红,爱打篮球的不知道“小皇帝”,简直都无法走入任何一个“肿瘤”社交圈啦!
除了----大数据,免费用,一不小心就能挖出一篇新文章这几个关键语句,想要进一步了解TCGA的朋友们继续往下看哦!不要停!
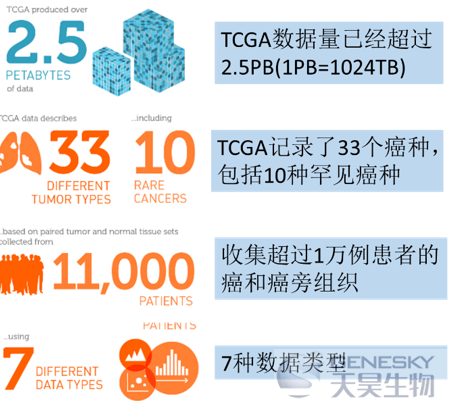
1、 看数字了解TCGA
2、 TCGA计划的使命与目标
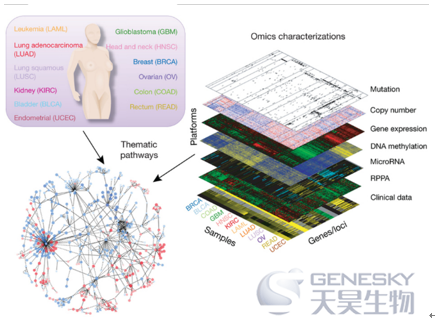
3 7种组学数据定义癌症基因组
4 TCGA与ICGC研究计划的关系
5 如何从TCGA数据中挖掘自己想要的信息?---先读读文献吧!
挖掘其实和“盗墓”有点像,如果这个“宝藏”现成的说明书都不读的话,又怎能“盗取”得到有用的宝藏呢!TCGA说明书,其实就是该计划开始以来TCGA团队发表的几十篇高分文章。几乎每篇文章,都是基于某个癌种几百例样本的多组学研究数据,详细展示了不同数据的分析方式和挖掘方法,并最终绘制某种癌症的基因组“图谱”。正是这样的文章不断地积累,才有了如今TCGA数据库中万例样本的多组学数据。
今天小编先和大家一起快速浏览一下2017年2月发表于《Cancer cell》杂志的一篇TCGA数据库文章:Integrated Molecular Characterization of Uterine Carcinosarcoma.
TCGA已发表的文章都可以在它的网页上找到(https://cancergenome.nih.gov/publications),建议大家重点阅读几篇后,再去构思自己的挖掘思路。
6 TCGA数据到底怎么挖掘呢?---在线数据库方式
目前已经有多个在线网站可以提供TCGA数据的分析与图形展示。其中最受欢迎的就是无需注册就能免费使用的cBioportal网站了(http://www.cbioportal.org)。
截至目前,cBioPortal网站已经整合了159个肿瘤基因组研究的数据,包括TCGA和ICGC等大型的肿瘤研究项目,涵盖了近三万例标本的数据,此外部分样品还包括了临床预后等表型的信息。除了提供数据下载,最重要的是cBioportal网站可以直接生成结果展示图,并且放入文章发表也没有问题。接下来,我们一起来试玩一下这个数据库,主页如下图:
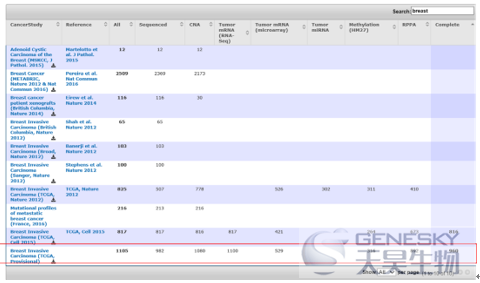
1) 数据库里乳腺癌研究有哪些,都有什么数据?
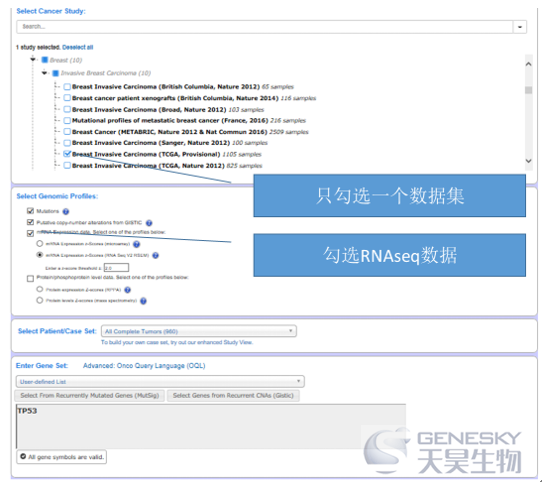
点击红框内的data sets,在搜索框输入breast(下图),可以看到网站里共记录了10个乳腺癌相关数据,并且后续的数字提示我们,不同的研究都包含了哪些分析平台数据,比如,红框内的研究具有最大样本量,而且数据信息看起来也是最全的。了解了这些数据的情况之后,我们就可以在主页面进行更有思路的筛选。
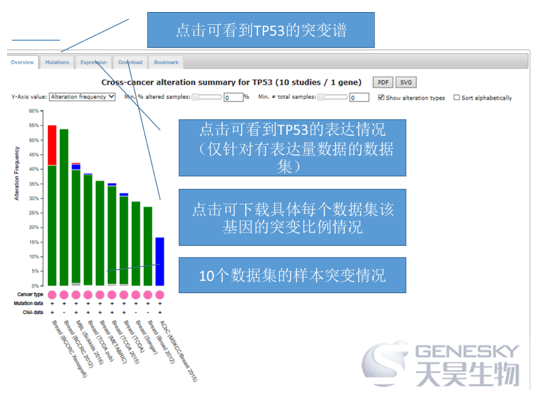
2) TP53在乳腺癌样本的突变情况如何?
在主页绿色的框内,选择Query,输入breast,就可以看到和1)里面一样的乳腺癌10个数据集。因为这10个数据集都有突变信息,所以我们可以全选(获得最大样本量),如下图,在基因列表区域输入TP53后点击submit。
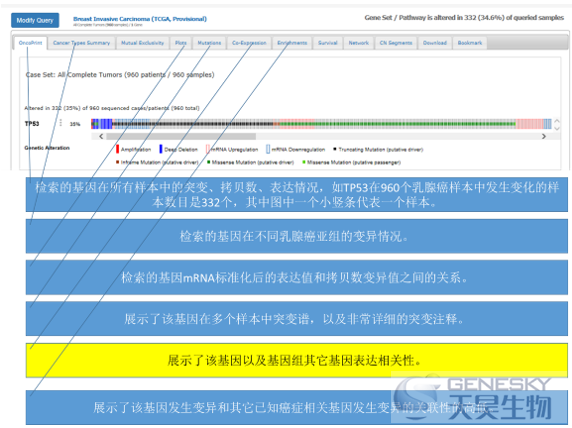
运行结束,首先展示的是10个数据集每一个的TP53变异样本比例,把鼠标放在bar图上,可以看到具体的比例数字。此外,还有mutation, expression, Download模块可以查看。其中,mutation模块点击后,可以看到所有变异的注释,有的还有蛋白3D结构注释,可以直接导出需要的图用于发表。
3) 如何查询TP53可能的下游基因?
如果我们想知道一个基因的下游分子是什么,通常需要在体外过表达或者敲除该基因,然后通过RT-PCR实验或者高通量测序的方法看哪些基因的表达量也发生了变化。cbioportal给出的Co-Expression分析模块,可以分析目标基因的表达量与基因组其他每个基因的表达相关系数,快速帮我们定位可能的下游基因。一般表达相关系数越大证明两者之间关系越密切,越有可能是上下游作用关系。因此,有了TCGA等大数据集的支持,可以进行有效的筛选,再开展功能试验。下面,我们就一起看看怎么寻找TP53可能的下游靶基因。首先,我们要重新勾选数据集,因为必须选择带有表达量信息的,通常是TCGA的数据集,才具有最全的信息,这里我们选择1105例样本的一个数据集。除了自动勾选的突变和CNV信息,我们需要手动勾选RNAseq来源的mRNA数据. 输入TP53后,submit。
由于我们选择的数据集分析平台很多,所以展示的结果如下图,有很多的模块。
大部分的模块在下图有进一步的解释。其中,标黄的说明,就对应了Co-Expression分析模块,展示了和TP53表达相关性非常高的基因。
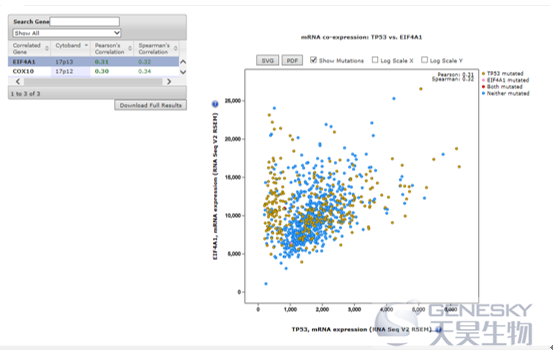
点击Co-Expression模块,下图显示了与TP53共表达的基因,以及Pearson和Spearman相关系数。点击基因,还可以绘制相关性散点图,每个点代表一个样本,并标识出哪些样本发生了突变。
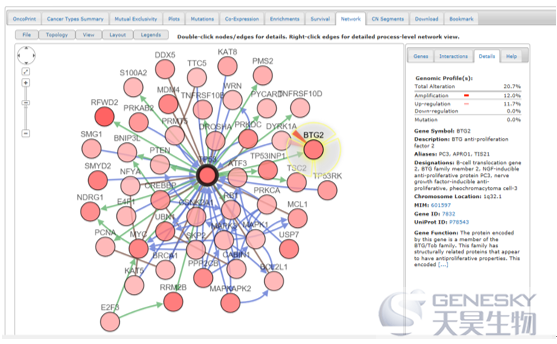
当然,有关TP53参与的网络或者信号通路,在Network模块里面会有更全面的展示,如下图。NetWork整合了来自Human ReferenceProtein Database(HPRD),Reactome,National Cancer Insititue (NCI)–Nature和The MemorialSloan-Kettering Cancer Center (MSKCC) Cancer Cell Map的数据进行网络图分析。鼠标左键双击每个节点,可以看到互做基因的更多信息。
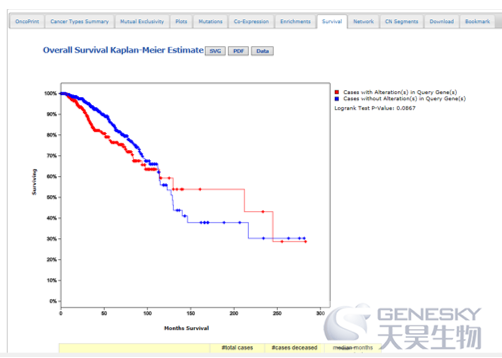
4)TP53的生存分析是什么样的?
点击survival模块,显示了TP53的生存曲线图,即有TP53变异的肿瘤样本和无变异的肿瘤样本之间的总的生存率预后曲线图和无病生存率预后曲线图。当然,能不能获得生存曲线图取决于数据集有没有临床信息。
以上的展示,尽管在突变层面能够比较详细直观的获得我们想要的信息,但是目前在线数据库对转录组数据,甲基化数据的分析展示还有非常有限的。因此,大部分的科研小能手,会通过cbioportal或者其它下载方式,获得TCGA数据的原始数据,从而进行更贴合自己研究内容的分析。
7 TCGA高级挖掘-----下载原始数据
为了追求更加原汁原味的TCGA数据,更好的开展挖掘,必须准备个小硬盘把原始数据下载下来才能放心。小编查了很多下载方法,可是试了10个,9个都不会。最终,只会R语言的小编,找到了RTCGAToolbox这个工具包,方便又好用。在线说明书链接在此 http://bioconductor.org/packages/release/bioc/manuals/RTCGAToolbox/man/RTCGAToolbox.pdf。
如果你不想去看这个英文说明,一看到命令行就头疼,但又想掌握如何用R语言下载TCGA原始数据的小伙伴,请关注“上海天昊生物”微信号。
如何用R语言下载TCGA原始数据,答案就在----TCGA数据库弄不临清,怎么好意思在肿瘤圈social?---下篇里!

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886